2022.9.12追記:PHENIX(現在のver.は1.20.1)のEnhanced maps -> Polder Mapsでpdbとmtzを読み込み、Omit selectionを入れて(例: chain a and resid 804)、適当なoutput prefixを入れて走らせた後、Maps -> Create a map from map coefficientsで、MTZ fileに先程のpolder mapの出力(XXX_polder_map_coeffs.mtz)、Model fileにpdbを読み込み、Atom selectionにomit selectionと同じものを記入し、適当にOutput file prefixを入れて走らせる。PHENIXのディレクトリの中のFFT_XX(job番号)というフォルダの中の、XXX_polder_map_coeffs_1.ccp4というファイル(2ではない)をPyMOL(今は2.4.2を使ってます)にドラッグアンドドロップ。isomeshにチェック入れて、levelを3-4くらいにして、bufferを2.0, carveにチェックを入れてloadすればマップが見えると思います。
2019.8.19追記:ここで紹介している方法よりも、PHENIXでpolder mapを描いた方が断然楽だし良い図が得られます。詳しくはofficialの紹介ページと論文をご見て下さい。
2016.09.29 Fushinobu
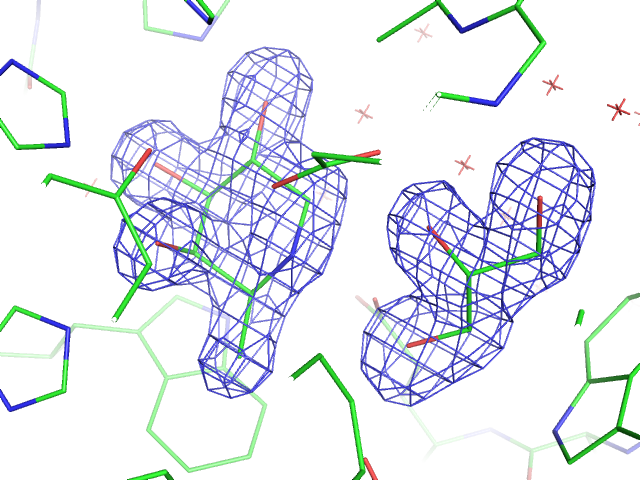
結合したリガンドの電子密度マップは論文にほぼ必ず載せる図ですね。一般的に用いられている通称「Fo -Fcオミットマップ」(リガンドをモデルから除いて描いたσA-weighted (mFo - DFc)exp(iφc) difference map)の作り方をここでご説明します。以前のバージョンだとweightがかかってなかったので(すみません)書き直してます。
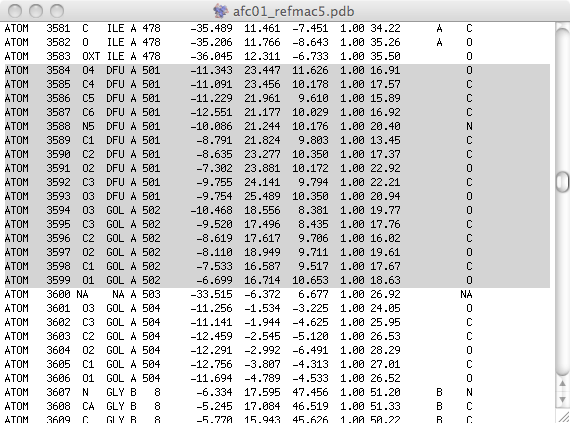
pdbファイル(例:afc01_refmac5.pdb)から表示したいリガンドの行だけを削除し(この場合、DFU A 501とGOL A 502)、afc01_refmac5noLIG.pdbなどとして保存する。注:ポリペプチド中のアミノ酸のマップを表示したい場合には、occupancy(B-factorの前の数字で通常は1.00になっている)をゼロにする、ということでいけるはず(未確認)

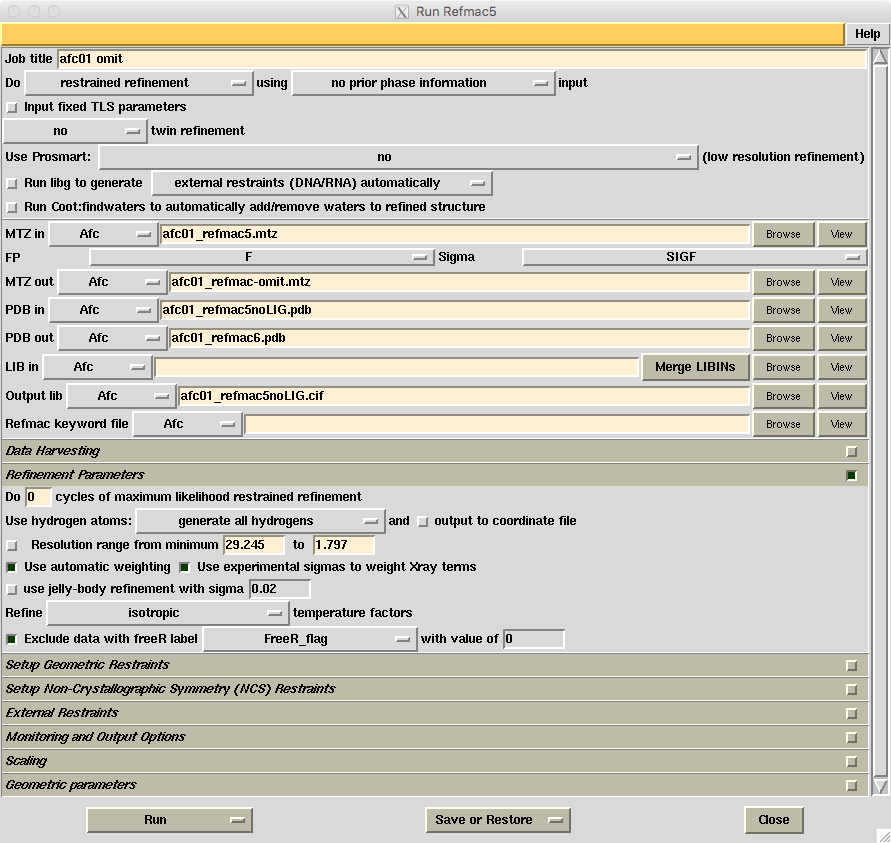
ccp4iで、Refinement→Run Refmac 5。MTZ inに最新のmtzファイル(afc01_refmac5.mtz)を、PDB inにさきほど作成したpdbファイル(afc01_refmac5noLIG.pdb)を入れる。出力されるmtzファイル(MTZ out)に、afc01_refmac-omit.mtzなどと任意の名前をつける。Refinement ParametersでDo 0 cycles ..にする。
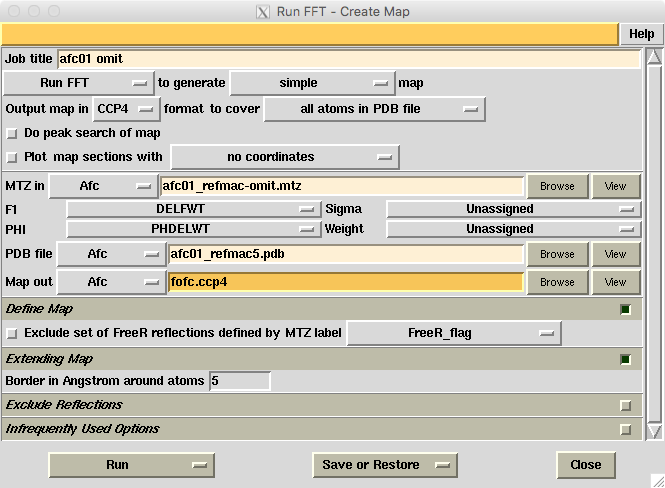
Map & Mask Utilities→Run FFT - Create Map。to generateのタブはsimple (map)で。to coverでall atoms in PDB fileを選ぶ。MTZ inに、先ほどRefmacが出力したmtzを選び、F1にDELFWTを、PHIにPHDELWTを選ぶ。PDB fileに、noLIGにする前のpdbファイル(afc01_refmac5.pdb)を、Map outは、***.mapとかになっていると思うが、書き換えて、短い名前で拡張子をccp4にする(fofc.ccp4など)。これは後でPyMolで読み込むため。Map outの出力がデフォルトではTEMPORARYになっていると思うので、それがイヤなら適当なフォルダに変更する(図ではccp4の作業ディレクトリAfcにしている)
PyMolを起動、noLIGにする前のpdbファイル(afc01_refmac5.pdb)を読み込む。File→Openでfofc.ccp4を読み込む。右の柱にfofcというオブジェクト(map)が現れる。fofcのAのところをクリック→mesh→@ level 3.0で、メッシュオブジェクト(fofc_mesh)が現れる。
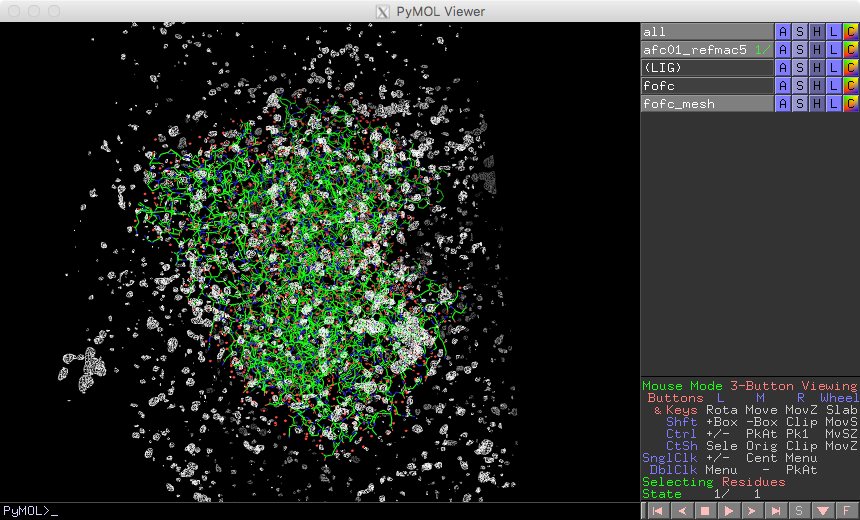
リガンド(この場合DFU A 501とGOL A 502)をselectしてLIGというselectionを作る。
>select LIG, (resn DFU and resi 501 and chain A) or (resn GOL and resi 502 and chain A)
右の柱の(LIG)のA→zoomで動く。LIGの周り1.0Å(かつ原子の周囲2.0Å)で4.0σのマップを表示するには、以下のように入力する。
>isomesh fofc_mesh, fofc, 4.0, LIG, 1, 0, 2
meshの太さは0.5か0.3くらいに(set mesh_width=0.5)、色はtv_blueかoliveがおすすめ(fofc_meshの右のC→blues→tv_blue)。その後は以下のようにして出力。
>bg_color white (背景を白にする)
>set ray_shadow=off (影を映さない)
>ray (レイトレース)
>png fofc (fofc.pngというファイルを書き出す)